IPSS-M: Molecular International Prognostic Scoring System
Seit 2012 verwenden wir bei myelodysplastischen Neoplasien (MDS) zur prognostischen Einteilung den IPSS-R. Im letzten Jahrzehnt haben große Studien gezeigt, dass auch die Molekulargenetik eine entscheidende Rolle dabei spielt (Bejar et al. NEJM 2011, Papaemmanuil et al. Blood 2013, Haferlach et al. Leukemia 2014). Dies spiegelt sich im Molecular International Prognostic Scoring System (IPSS-M) wieder, der neben Zytogenetik, Blutbild und Blastenzahl auch Mutationen berücksichtigt (Bernard et al. NEJM Evidence 2022).
Was steckt hinter dem IPSS-M?
Ein internationales Forschungsteam (The International Working Group for the Prognosis of MDS) untersuchte 2.957 MDS Patient:innen und erarbeitete eine verbesserte Prognoseabschätzung basierend auf folgenden Werten:
- Hämoglobin
- Thrombozytenzahl
- Blastenzahl (KM)
- zytogenetische Prognose-Kategorie (wie im IPSS-R)
- molekulargenetische Informationen zu 31 Genen
Besonders heben die Autor:innen die Bedeutung von FLT3 Veränderungen (TKD oder ITD) sowie von KMT2A-PTD (MLLPTD) hervor. Diese sind im MDS sehr selten, haben aber einen stark negativen Einfluss auf die Prognose. Bei TP53 spielt der Status „multihit“ eine besondere Rolle; darunter versteht man das Vorliegen von zwei oder mehr Mutationen, einer Mutation und Deletion (del(17p)) oder einer Mutation mit einem kopien-neutralen Verlust der Heterozygotie (loss of heterozygosity; cnLOH). Auch zu SF3B1 liegen neue Erkenntnisse vor. Die positive prognostische Bedeutung verliert sich, wenn gleichzeitig eine del(5q) oder eine Mutation in einem der folgenden Gene vorliegt: BCOR, BCORL1, NRAS, RUNX1, SRSF2 oder STAG2.
Wie lässt sich der IPSS-M berechnen?
Neben dem Blutbild benötigt man:
- Zytomorphologie aus dem Knochenmark für den Blastenanteil
- Zytogenetik/Chromosomenanalyse
- das IPSS-M-Panel nach Bernard et al. in
der Molekulargenetik
Das Ergebnis des IPSS-M ist ein Zahlenwert, welcher in sechs zugeordnete Risiko-Kategorien eingeteilt wird (s. Abb. 1):
- Very Low (VL)
- Low (L)
- Moderate Low (ML)
- Moderate High (MH)
- High (H)
- Very High (VH)
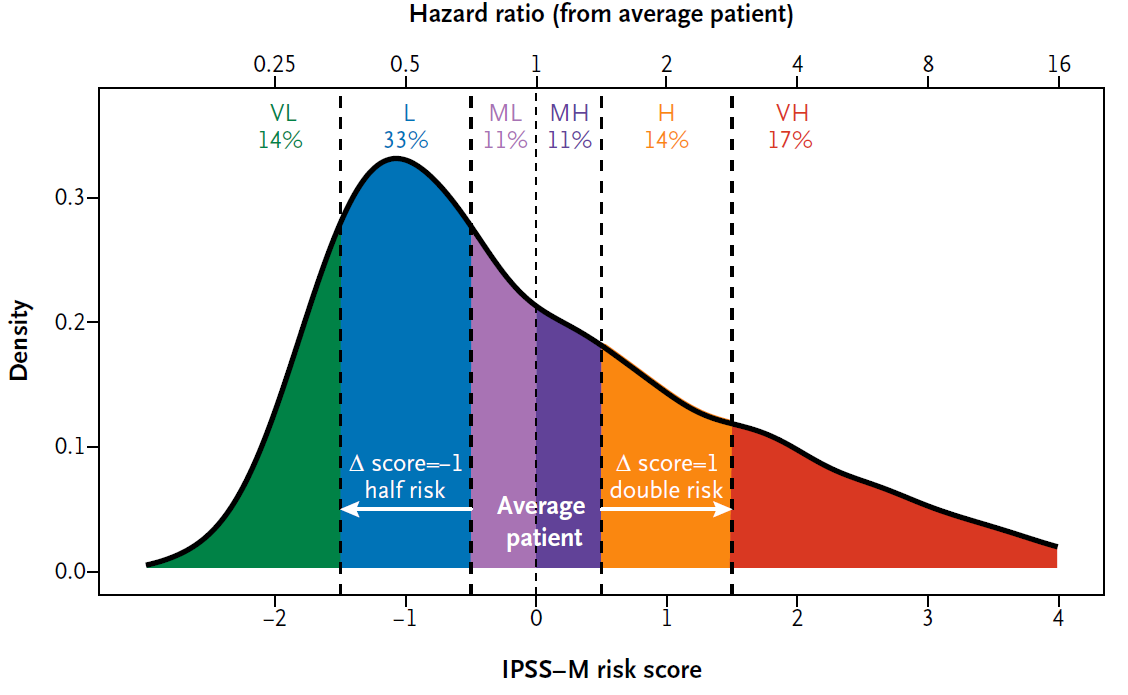
Die Risikogruppen zeigen in der Publikation eine
starke prognostische Separation bei Gesamtüberleben, leukämiefreiem Überleben
und AML-Transformation.
- Die Berechnung des IPSS-M kann auf der Webseite der MDS Foundation erfolgen. Zur Bedienung ein paar praktische Tipps:
- Am Ende des Befunds finden Sie die Tabelle Durchgeführte Analysen. Eine Mutation vom Typ Tier 1 und 2 geben Sie als „Mutated“ ein.
- Im WebTool wird der ursprüngliche Genname MLL verwendet. Wir verwenden die aktuelle Bezeichnung KMT2A.
- TP53: Wie
viele Mutationen vorliegen, können Sie in der Tabelle Veränderungen erkennen. Zusätzlich muss die „Maximum TP53 VAF“ eingegeben werden. Diese entnimmt man
der Spalte VAF in selbiger Tabelle.
Zukünftig werden wir auch die Information zum „Loss of heterozygosity“
bereitstellen.
Wir arbeiten aber aktuell auch an einer entsprechenden Anpassung unserer Befundung. Wenn Sie uns Blutbildwerte übermitteln und eine zytomorphologische, zytogenetische und molekulargenetische Diagnostik beauftragen, werden wir Ihnen in Kürze auch den IPSS-M im Rahmen unseres Integrierten Befundes bereitstellen können.
Die Autorin

»Sie haben Fragen zum Artikel oder wünschen weitere Informationen zum IPSS-M? Schreiben Sie mir gerne eine E-Mail.«
Dr. rer. nat. Constance Bär