Jede Zelle zählt - Etablierung der Einzelzellsequenzierung am MLL
Die Erstellung von molekulargenetischen Profilen hämatologischer Neoplasien hat wichtige Erkenntnisse über die molekulare Pathogenese von Blutkrebs geliefert und ist ein essentieller Bestandteil in der klinischen Diagnostik. Die detaillierte Auflösung der genetischen Landschaft von Leukämien und Lymphomen hat zur Entwicklung neuer zielgerichteter Therapien geführt und den Weg zur Präzisionsmedizin geebnet. Jedoch kann die Analyse von Gruppen von Zellen wichtige Aspekte der intra-tumoralen Heterogenität verschleiern, die der klonalen Evolution und dem Ansprechen auf die Behandlung zugrunde liegen. Zu diesem Zweck ist die Einzelzellsequenzierung vielversprechend, denn sie ermöglicht die Untersuchung der Genexpression, der epigenetischen Heterogenität und der subklonalen DNA-Architektur mit einer Auflösung, die mit der Massensequenzierung nicht erreicht werden kann.
Für die Untersuchung bestimmter Modalitäten in einzelnen Zellen stehen
mehrere Techniken zur Verfügung, darunter die Analyse des Transkriptoms, des
Genoms, der Chromatin-Zugänglichkeit und der DNA-Methylierung. Am MLL haben wir
uns aktuell auf die gepaarte Analyse des Transkriptom- und Epigenomprofils für
ein tieferes Verständnis der Genregulation fokussiert und den 10X Multiome ATAC
+ Genexpressionsassay für Forschungsprojekte etabliert. Für diesen Assay werden
Zellkerne isoliert, markiert/kodiert und dann DNA und RNA für die Sequenzierung
vorbereitet.
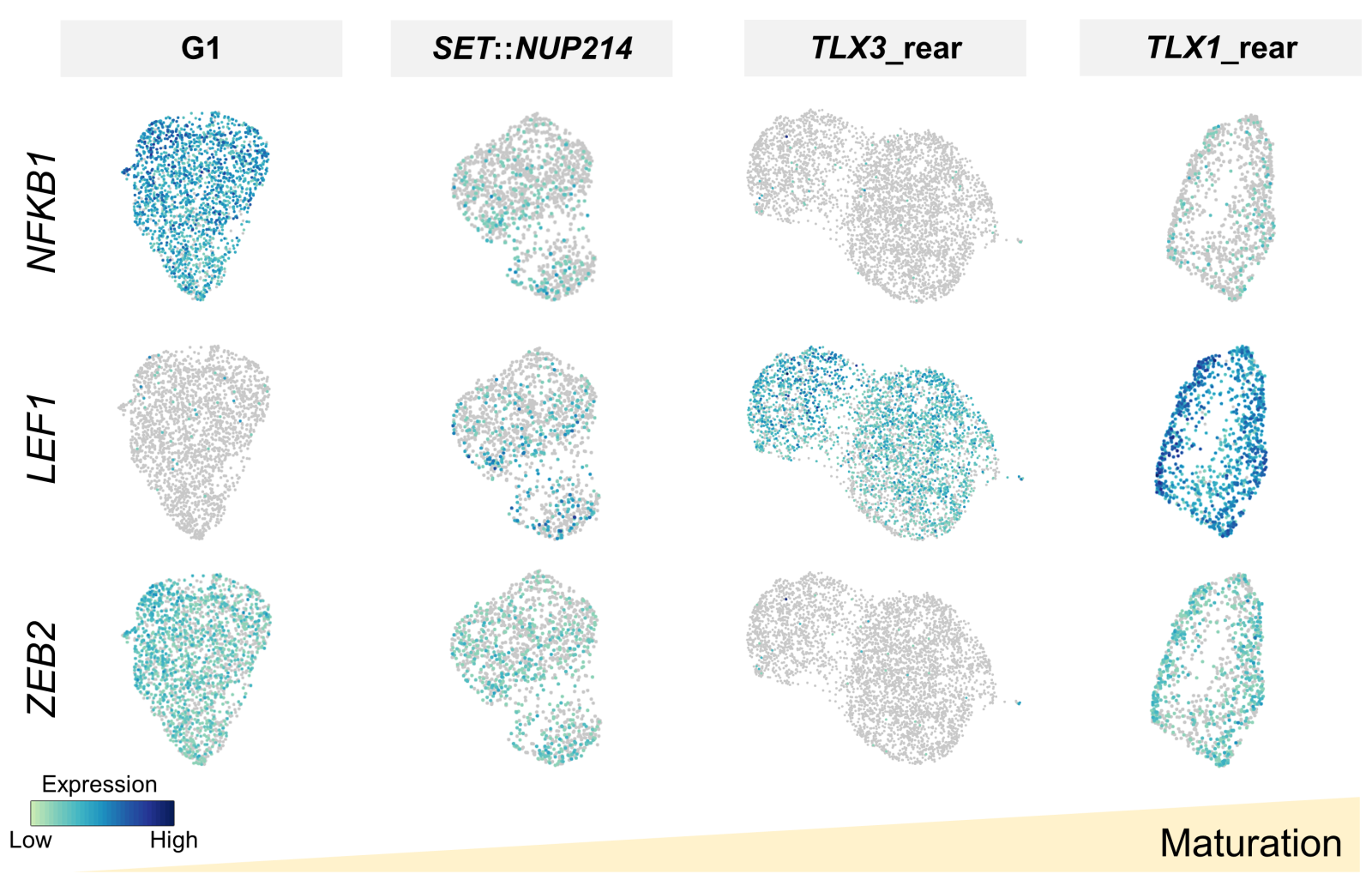
In einem ersten Projekt haben wir uns mit der Analyse der T-Zell akuten lymphoblastischen Leukämie (T-ALL) beschäftigt. Bei der T-ALL geht man davon aus, dass die unterschiedlichen genetischen Subgruppen verschiedene Ursprungszellen haben. Unsere Einzelzell-Multiomanalyse bestätigte die unterschiedlichen T-ALL Reifungsstadien durch die gruppenabhängige Expression diverser Markergene (NFKB1, LEF1 und ZEB2, Abb. 1) und die Aktivierung unterschiedlicher Regulationsprogramme. Zusätzlich beschäftigen wir uns im Zuge des innovativen Ausbildungsnetzwerkes „INTERCEPT-MDS“ mit der Identifizierung von Biomarkern auf Einzelzellebene, welche eine frühzeitige Erkennung und potenzielle Behandlung von klonalen myeloischen Erkrankungen ermöglichen können.
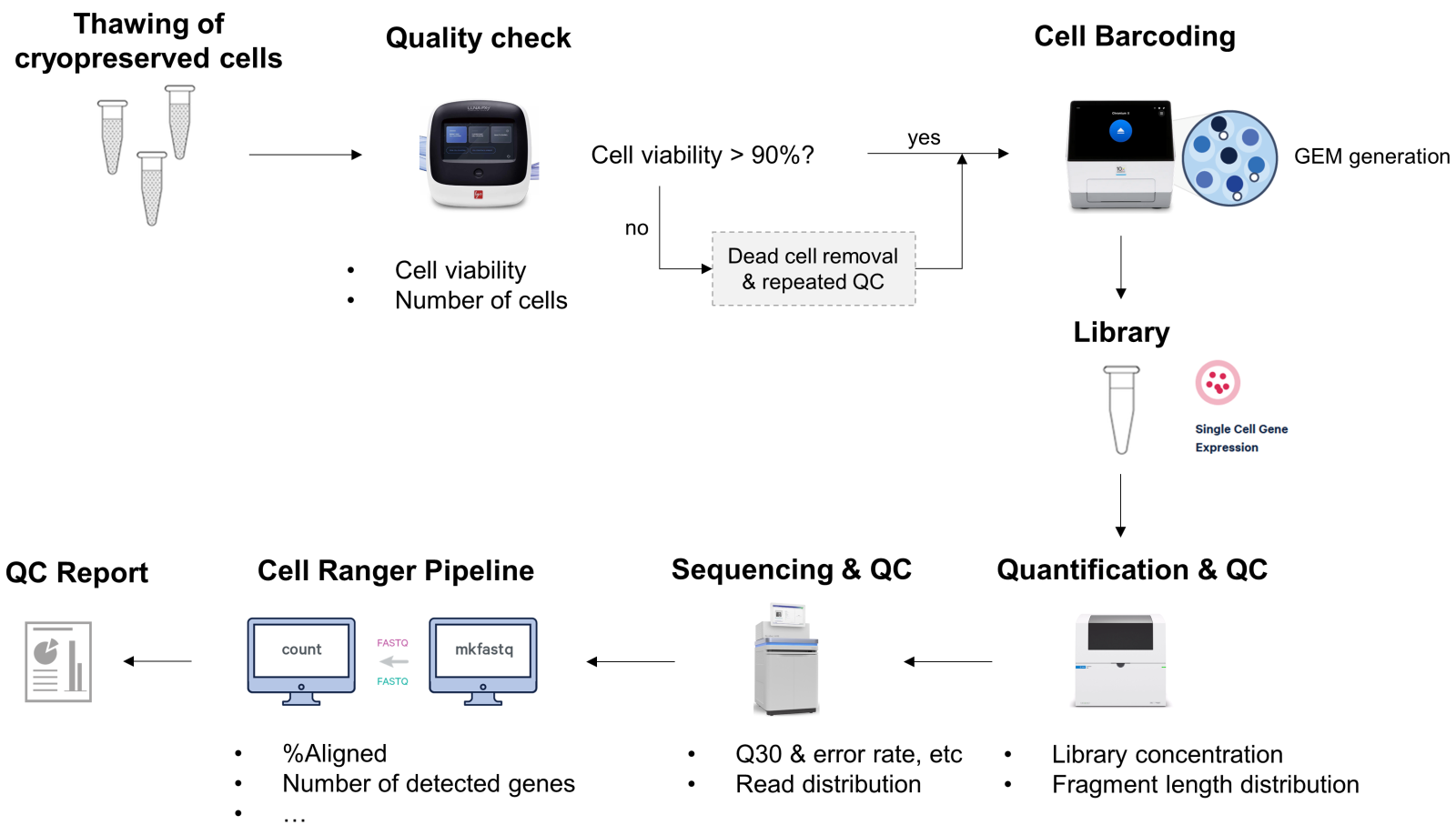
Wir haben auch das Portfolio unseres Sequenzierservices MLLSEQ für die Einzelzellanalyse mit dem Single Cell 3‘ Genexpressionsassay erweitert. Die Einzellzellsequenzierung erfordert besonders hochqualitatives Eingangsmaterial (1-2 Mio. Zellen, Zellviabilität > 90%) und unterliegt strikten Qualitätskontrollen (Abb. 2). So sind wir von der Probenvorbereitung über die library preparation bis hin zur Datenverarbeitung in den Best Practices geschult und stolz, dass MLLSEQ Teil des 10x Genomics Service Provider Programs ist.
Auch wenn die Einzelzellsequenzierung aktuell noch keine direkte diagnostische Relevanz hat, birgt das Feld ein großes Potenzial nicht nur für die Grundlagenforschung, sondern auch für klinische Ansätze (z.B. MRD Monitoring), mit denen wir uns ebenfalls in Zukunft beschäftigen werden – wir halten Sie auf dem Laufenden!

»Sie haben Fragen zum Artikel oder wünschen weitere Informationen? Schreiben Sie mir gerne eine E-Mail.«
Dr. Wencke Walter