Every cell counts - establishing single cell sequencing at the MLL
Molecular genetic profiling of hematologic neoplasms has provided important insights into the molecular pathogenesis of blood cancers and is an essential component of clinical diagnostics. The detailed resolution of the genetic landscape of leukemias and lymphomas has led to the development of new targeted therapies and paved the way for precision medicine. However, analysis of groups of cells can obscure important aspects of intra-tumor heterogeneity underlying clonal evolution and response to treatment. To this end, single-cell sequencing is promising because it allows the study of gene expression, epigenetic heterogeneity and subclonal DNA architecture at a resolution that cannot be achieved with bulk sequencing.
Several techniques are available to study specific modalities in single cells, including analysis of the transcriptome, genome, chromatin accessibility and DNA methylation. At MLL, we have currently focused on paired analysis of the transcriptome and epigenome profile for a deeper understanding of gene regulation and have established the 10X Multiome ATAC + gene expression assay for research projects. For this assay, cell nuclei are isolated, labeled/encoded and then DNA and RNA are prepared for sequencing.
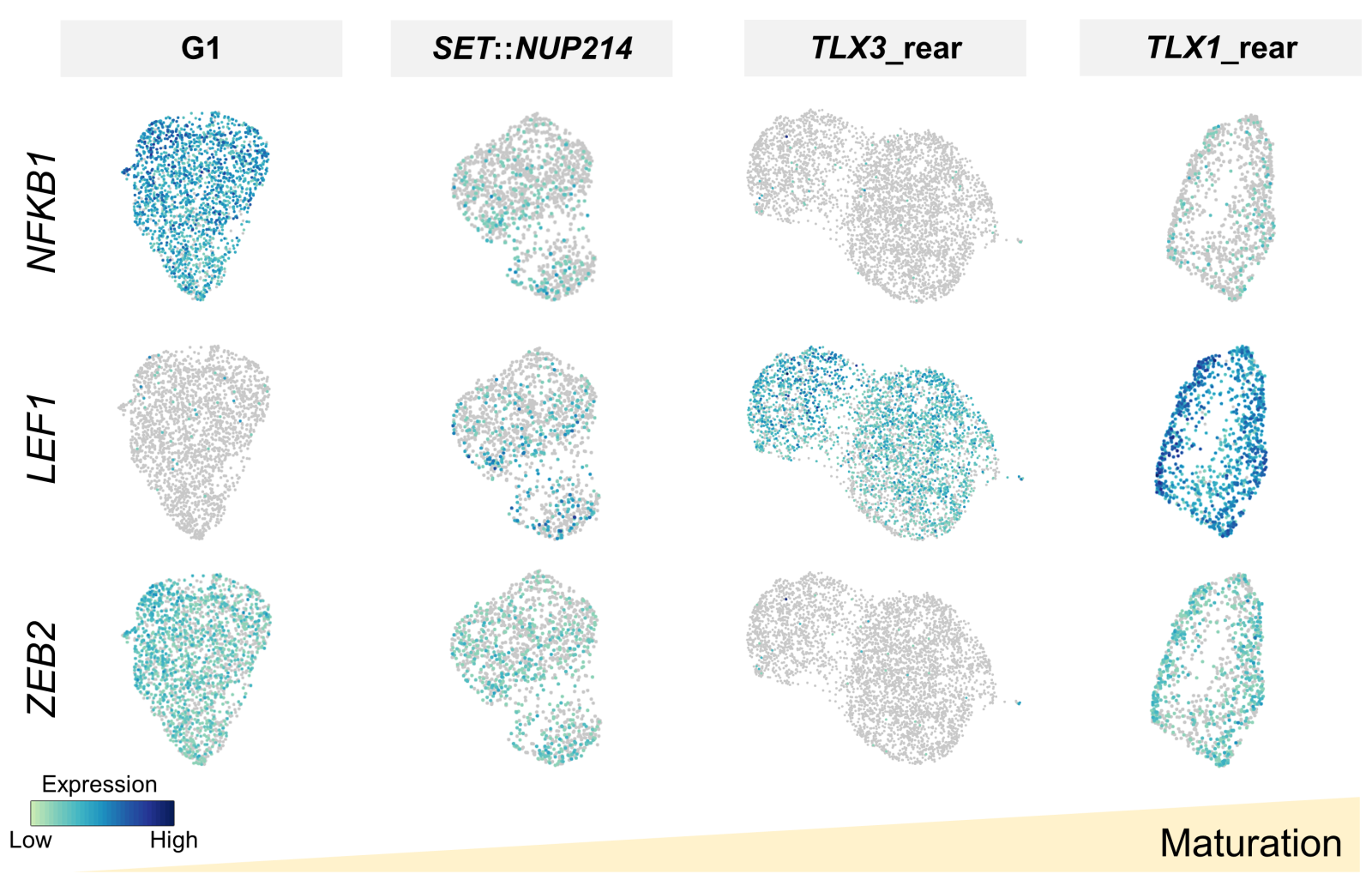
In a first project, we worked on the analysis of T-cell acute lymphoblastic leukemia (T-ALL). In T-ALL, it is assumed that the different genetic subgroups have different cells of origin. Our single-cell multi-omics analysis confirmed the different T-ALL maturation stages by the group-dependent expression of various marker genes (NFKB1, LEF1 and ZEB2, Fig. 1) and the activation of different regulatory programs. In addition, as part of the innovative "INTERCEPT-MDS" training network, we are working on the identification of biomarkers at the single-cell level, which could enable early detection and potential treatment of clonal myeloid diseases.
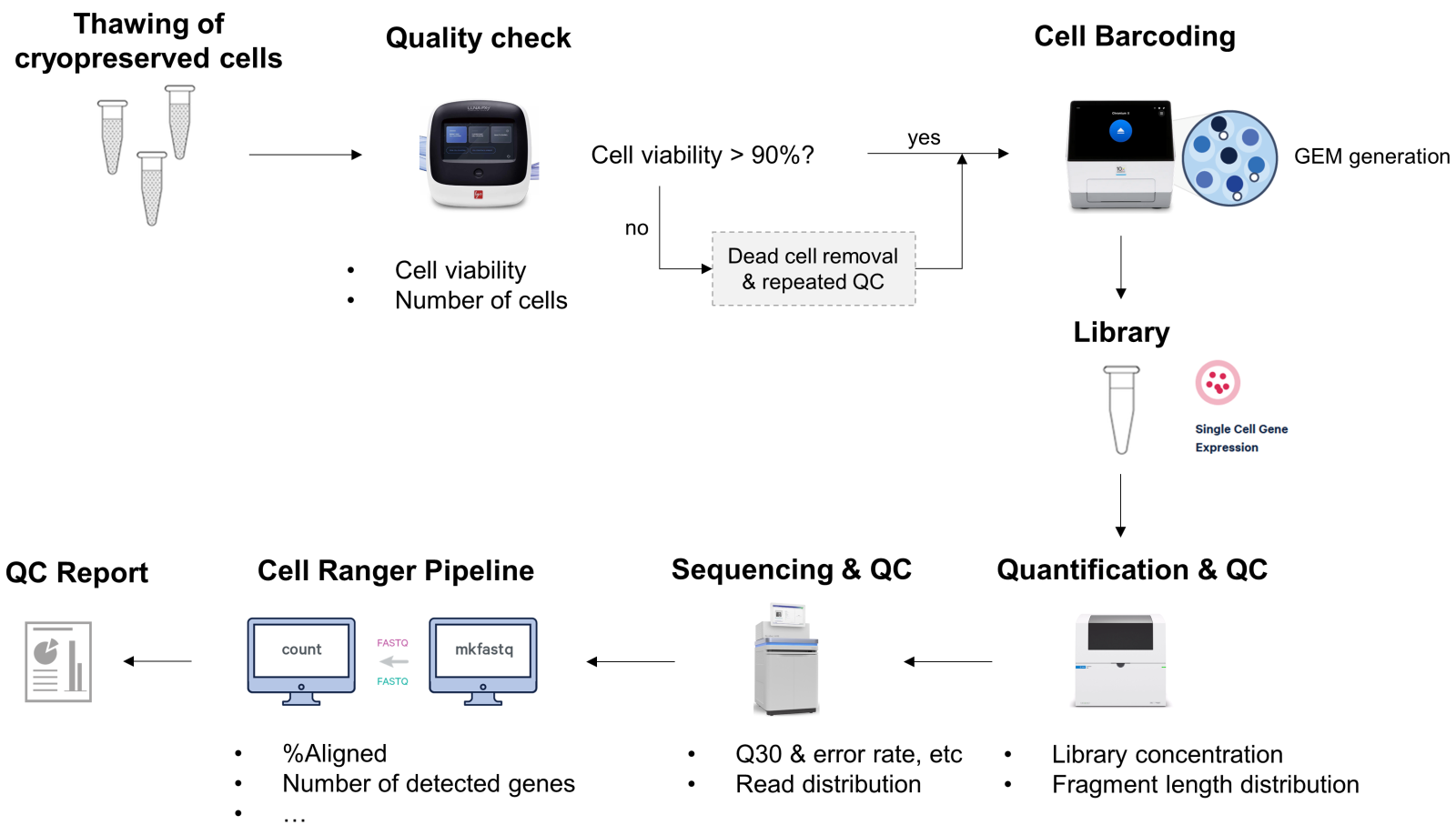
Wir haben auch das Portfolio unseres Sequenzierservices MLLSEQ für die Einzelzellanalyse mit dem Single Cell 3‘ Genexpressionsassay erweitert. Die Einzellzellsequenzierung erfordert besonders hochqualitatives Eingangsmaterial (1-2 Mio. Zellen, Zellviabilität > 90%) und unterliegt strikten Qualitätskontrollen (Abb. 2). So sind wir von der Probenvorbereitung über die library preparation bis hin zur Datenverarbeitung in den Best Practices geschult und stolz, dass MLLSEQ Teil des 10x Genomics Service Provider Programs ist.
Auch wenn die Einzelzellsequenzierung aktuell noch keine direkte diagnostische Relevanz hat, birgt das Feld ein großes Potenzial nicht nur für die Grundlagenforschung, sondern auch für klinische Ansätze (z.B. MRD Monitoring), mit denen wir uns ebenfalls in Zukunft beschäftigen werden – wir halten Sie auf dem Laufenden!

»Sie haben Fragen zum Artikel oder wünschen weitere Informationen? Schreiben Sie mir gerne eine E-Mail.«
Dr. Wencke Walter