Chromosomenanalyse 5.0 – Automatisierung, Digitalisierung und künstliche Intelligenz
Die Methode
Die Chromosomenanalyse gilt als arbeitsintensive und anspruchsvolle Methode, deren Durchführung und Ergebnisinterpretation hohe Anforderungen an die Fähigkeiten des Personals stellt.
Es werden vitale teilungsfähige Zellen benötigt, die ein bis drei Tage kultiviert werden, mit anschließender Präparation der Metaphasen und Färbung der Chromosomen mittels spezifischer Bänderungstechnik. Die sogenannte Giemsa-Bänderung verleiht dabei jedem Chromosom ein spezifisches Bandenmuster, welches eine eindeutige Identifizierung der Chromosomen sowie die Detektion von Chromosomenveränderungen ermöglicht. Die Metaphasen werden computergestützt in einem automatisierten System am Mikroskop aufgenommen und anschließend für das Erstellen der Karyogramme digital bearbeitet. Basierend auf der Analyse von mindestens 20 Karyogrammen wird eine Karyotypformel abgeleitet und ein zytogenetischer Befund erstellt.
Reduktion der Bearbeitungszeit durch KI
Im MLL werden bereits seit vielen Jahren automatisierte Verfahren – von der vollautomatisierten Aufarbeitung der Zellen bis hin zur Präparation der Metaphasen sowie Färbe-, Medium- und Auftropfroboter – im Labor der Chromosomenanalyse eingesetzt und stetig weiterentwickelt. In Zusammenarbeit mit der Firma MetaSystems hat das MLL zudem sogenannte DNNs (deep neural networks) für Objekterkennung, Qualitätsbeurteilung und Klassifikation menschlicher Chromosomen trainiert. Seit Ende des Jahres 2019 werden solche KI-basierten Algorithmen routinemäßig in der Chromosomenanalyse eingesetzt und fortlaufend weiterentwickelt. So kann ein Karyogramm inzwischen voll automatisiert erstellt werden und manuelle Bildbearbeitungsschritte entfallen komplett.
- Dauer für manuelles Erstellen eines Karyogramms: ca. 2 bis 3 Minuten
- Dauer für Erstellen eines Karyogramms mittels KI (inklusive anschließender Kontrolle durch einen Zytogenetiker): ca. 25 Sekunden
Wie das in der Praxis aussieht, haben wir in einem Video für Sie zusammengefasst. Hier geht’s zum Film.
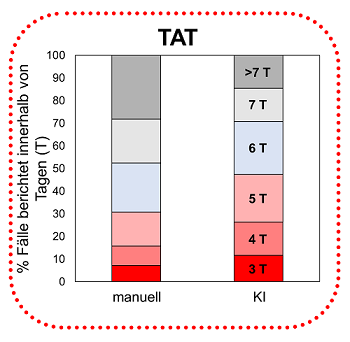
Durch den Einsatz von KI konnte die Bearbeitungszeit (turnaround time, TAT) in der Chromosomenanalyse deutlich reduziert werden. So können mittlerweile bei fast 50 % der Fälle die Ergebnisse innerhalb von ≤5 Tagen berichtet werden im Vergleich zu ca. 30 % der Fälle vor Einsatz der KI. Entsprechend konnte der Anteil an Fällen, deren Bearbeitungszeit länger als 7 Tage beträgt, von 28 % auf 15 % gesenkt werden.
Eine KI-basierte Qualitätsbeurteilung der Metaphasen ermöglicht zudem eine breite Analyse über unterschiedliche Qualitätsstufen hinweg und gewährleistet somit eine objektive Beurteilung des gesamten Falls. Darüber hinaus erleichtert der Einsatz von KI die gezielte Suche nach Metaphasen mit aberrantem Karyotyp, wodurch insbesondere kleine aberrante Klone besser erfasst werden können.
Relevanz für die Leukämiediagnostik
Auch in Anbetracht des raschen Fortschritts im Bereich der Ganzgenomsequenzierung bleibt die Chromosomenanalyse ein essentieller Bestandteil der Diagnostik hämatologischer Neoplasien. Die Relevanz der Chromosomenanalyse zeigt sich unter anderem in folgenden Aspekten:
- Kosteneffizienz
- In manchen Fällen einzige Möglichkeit der Detektion von kleinen Klonen durch Proliferationsvorteil während der Kultivierung
- Einzelzellanalyse:
- Unterscheidung von unabhängigen Klonen, z.B. zwischen Klonen einer myeloischen und gleichzeitig vorliegenden lymphatischen Erkrankung
- Unterscheidung zwischen klonaler Evolution (Weiterentwicklung eines Klons) und potenzieller hämatologischer Zweiterkrankung in einem unabhängigen Klon - Karyotyp als unabhängiger prognostischer Faktor bei vielen hämatologischen Neoplasien (z.B. IPSS-R bei MDS oder komplexer Karyotyp bei CLL)
- Karyotyp in manchen Fällen therapieentscheidend (z.B. bei AML mit Myelodysplasie-assoziierten Veränderungen)
Durch Automatisierung und den Einsatz von KI hat sich die als aufwändig geltende Chromosomenanalyse zu einer schnelleren und sensitiveren Methode entwickelt, die nach wie vor bei vielen hämatologischen Neoplasien von hoher Bedeutung ist. Zur Orientierung, in welchen Fällen eine Chromosomenanalyse indiziert ist, finden Sie auf unserer Website Empfehlungen zu dem methodischen Spektrum für die jeweilige Fragestellung bzw. Verdachtsdiagnose.
Die Autorinnen

»Sie haben Fragen zum Artikel oder wünschen weitere Informationen? Schreiben Sie mir gerne eine E-Mail.«
Dr. rer. nat. Isolde Summerer
Diplombiologin
Bereichsleitung Zytogenetik
Stellv. Abteilungsleitung Chromosomenanalyse-Befundung
Stellv. Abteilungsleitung FISH-Befundung
isolde.summerer@mll.com