Myelodysplastische Neoplasie (MDS)
- Methode:
- Antikoagulans:
- Empfehlung:
- Methode:Zytomorphologie
- Antikoagulans:EDTA
- Empfehlung:obligat
- Methode:Immunphänotypisierung
- Antikoagulans:EDTA oder Heparin
- Empfehlung:fakultativ
- Methode:Chromosomenanalyse
- Antikoagulans:Heparin
- Empfehlung:obligat
- Methode:FISH
- Antikoagulans:EDTA oder Heparin
- Empfehlung:fakultativ
- Methode:Molekulargenetik
- Antikoagulans:EDTA oder Heparin
- Empfehlung:obligat
Auf Basis der aktuellen Leitlinien und des aktuellen Forschungsstandes ergeben sich verschiedene diagnostische Empfehlungen für Patienten mit myelodysplastischer Neoplasie. Wir haben Ihnen die wichtigsten Infos zur Klassifikation und den diagnostischen Methoden am MLL zusammengefasst. Zudem haben wir weiterführende Links zur Prognose und Therapie bei myelodysplastischer Neoplasie zusammengestellt, damit Sie sich tiefergehend informieren können.
Klassifikation bei MDS
Myelodysplastische Neoplasien (MDS, früher: Myelodysplastische Syndrome) sind erworbene klonale Knochenmarkerkrankungen, die bevorzugt im höheren Lebensalter auftreten. Die MDS ist geprägt von einer proliferativen und apoptotischen Pathologie der hämatopoetischen Vorläuferzellen. Zugleich liegt häufig ein inflammatorisches Mikromilieu der hämatopoetischen Stammzellnische vor, verbunden mit einer zunehmenden Genominstabilität (Stubbins et al. 2022). Folge ist oft eine Anämie und auch Neutropenie und/oder Thrombozytopenie. Dysplasiezeichen sind in mindestens einer der drei hämatopoetischen Zelllinien zu erkennen, und es finden sich gehäuft leukämische Transformationen in eine AML. Die genetische Charakterisierung spielt eine zunehmend große Rolle in der Diagnostik, was auch in der 2022 neu erschienenen WHO-Klassifikation abgebildet wird. In dieser werden nun „MDS mit definierenden genetischen Veränderungen“ und „MDS, morphologisch definiert“ unterschieden (WHO 2022):

Die neue Aufteilung der Subgruppen soll eine genauere Klassifikation und somit die Grundlage für zielgerichtete Therapien schaffen. Da die Anzahl der dysplastischen Zelllinien in der Regel dynamisch ist und nicht unbedingt einen spezifischen MDS-Typ definiert, ist die Unterscheidung zwischen Ein- und Mehrliniendysplasie nach WHO nun optional. Innerhalb der „MDS, morphologisch definiert“ stellt zudem das „MDS, hypoplastisch (MDS-h)“ eine neue definierte Subgruppe mit Zytopenie, Dysplasie und stark verringerter Knochenmarkzellularität dar. Die Subgruppe der „MDS, nicht klassifizierbar“ wird in der neuen WHO-Klassifikation nicht mehr verwendet.
MDS: Diagnostische Methoden und ihre Bedeutung
MDS: Prognose
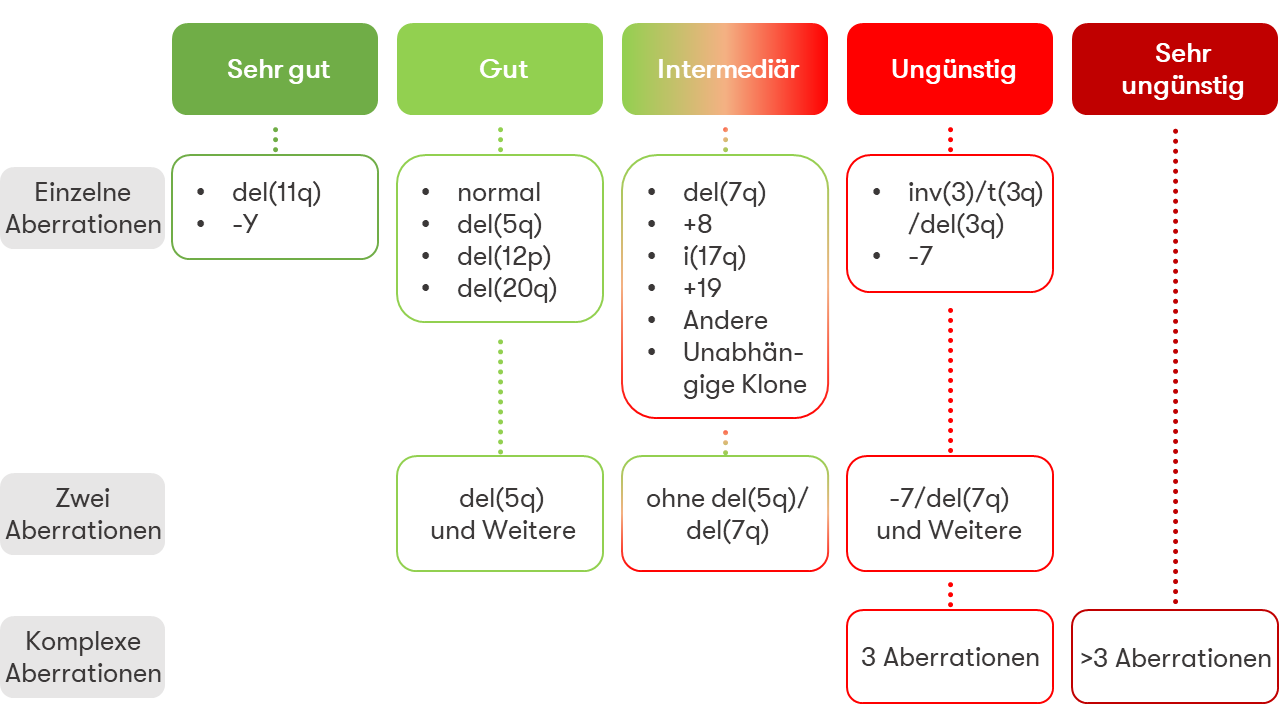
Wesentlicher Pfeiler der Prognoseeinstufung für Patienten mit MDS war für viele Jahre das "International Prognostic Scoring System" (IPSS) (Greenberg et al. 1997). Für eine verbesserte bzw. detailliertere Risikostratifizierung der Patienten mit MDS wurde das IPSS im Jahr 2012 überarbeitet (Revised-IPSS, IPSS-R) (Greenberg et al. 2012). Die inzwischen starke Bedeutung der Molekulargenetik wird in einem neuen Prognose-Score (IPSS-M) deutlich, in dem zusätzlich zu klinischen und zytogenetischen vor allem molekulargenetische Befunde berücksichtigt werden. Die Prognoseabschätzung basiert in diesem neuen System auf dem Hämoglobinwert, der Thrombozytenzahl, der Blastenzahl im Knochenmark, der zytogenetischen Prognose-Kategorie (wie im IPSS-R) und molekulargenetischen Informationen zu 31 Genen. Eine Zusammenfassung der Informationen finden Sie in einem Übersichtsartikel auf unserer Website (Bernard et al. 2022). Hier gelangen Sie zur Prognoseberechnung des IPSS-Scores, des IPSS-R-Scores, WPSS-Scores und des neuen IPSS-M unter Berücksichtigung der molekulargenetischen Ergebnisse.
MDS: Empfehlung & Therapie
Die zytomorphologische Knochenmarkdiagnostik in Kombination mit der Zytogenetik und der Molekulargenetik stellen den aktuellen Goldstandard in der MDS-Diagnostik dar (Bernard et al. 2022). Aktuelle Studien können über das MDS-Patientenportal und die Deutsche MDS-Studiengruppe eingesehen werden.
Da MDS dynamische Erkrankungen sind, spricht sich die Onkopedia-Leitlinie für ein jährliches Screening in den Bereichen Zytomorphologie, Zytogenetik, FISH und der Molekulargenetik aus - insbesondere, wenn im individuellen Fall zu erwarten ist, dass sich eine Risikostratifikation verändern kann (Onkopedia Leitlinie MDS 2023).
Die Therapie einer myelodysplastischen Neoplasie nach deutscher Leitlinie erfolgt u.a. in Abhängigkeit der Risikogruppe sowie des Alters und des klinischen Zustands der Patienten (Onkopedia Leitlinie MDS).
Stand: September 2023