62. ASH Annual Meeting & Exposition – ein Nachbericht
Auch im Jahr 2020, diesmal als virtueller Kongress, war das MLL beim 62. Annual Meeting & Exposition der American Society of Hematology (ASH) gut durch eigene Beiträge und in Kooperationen vertreten. Insgesamt hat das MLL mit seinem Team vier Vorträge gehalten und 11 Poster inklusive Kurzvortrag vorgestellt.
Die Schwerpunkte dabei waren die genauere Charakterisierung und Klassifizierung von Leukämien und Lymphomen ebenso wie die Anwendung neuer Methoden bis hin zu Genom-Sequenzierung und künstlicher Intelligenz, sowohl in Forschung als auch in der Routinediagnostik.
Insgesamt 12 Wissenschaftler aus dem MLL haben die Ergebnisse der Forschungsprojekte präsentieren dürfen. Als Vortrag aus dem Bereich der Zytomorphologie hat Herr Dr. C. Pohlkamp die aktuellen Ergebnisse vorgetragen, mit Hilfe von künstlicher Intelligenz (Kooperationen mit AWS) das periphere Blutbild zu differenzieren. Die dabei erreichten richtigen Voraussagen zur einzelnen Zellsorte benigner und maligner Zellen im peripheren Blut liegt mit dem getesteten Algorithmus bei 92%. Die dabei entwickelten Tools können Cloud basiert angewendet werden und befinden sich gerade in der prospektiven Testung (BELUGA-Studie) parallel zum Goldstandard der peripheren Blutbilddifferenzierung durch MTAs und Hämatologen.
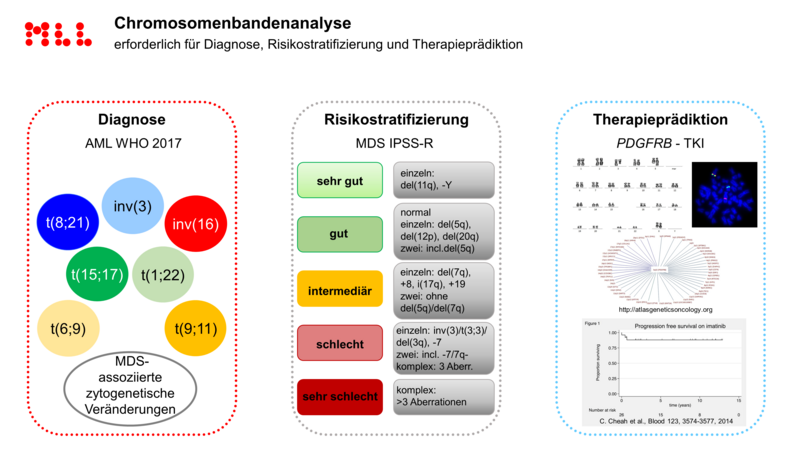
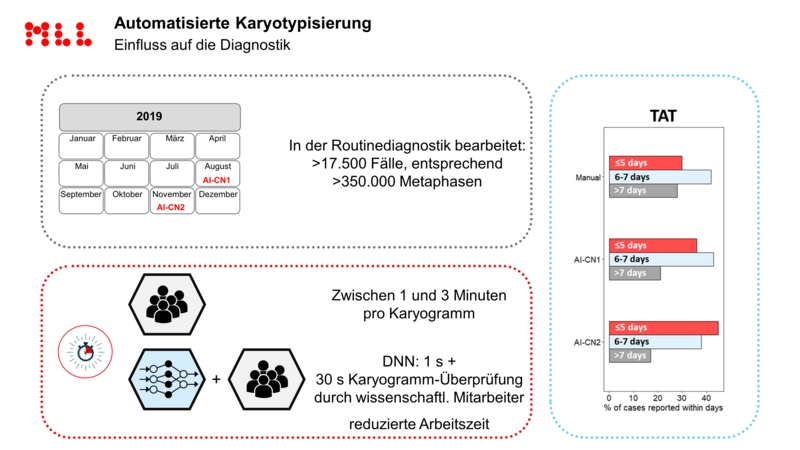
Für die klassische Chromosomenanalyse hat Frau Prof. C. Haferlach den aktuellen Stand der Dinge vorgestellt: Dabei ist es dem MLL gelungen, zusammen mit der Firma MetaSystems Algorithmen zu entwickeln, die bei der Karyotypisierung das Fachpersonal von MTAs und Biologen unterstützen können. Schon weit über 20.000 Patienten sind damit mit Unterstützung von Algorithmen der künstlichen Intelligenz in der Routinediagnostik prozessiert worden und führen zu einer deutlichen Verkürzung der Befundungszeit der Chromosomenanalyse zum Teil um zwei Tage im Vergleich zur vorherigen Dauer des Workflows.
In der Molekulargenetik spielt zunehmend die Panel-Sequenzierung mit Next-Generation-Sequencing (NGS) eine Rolle. Speziell auch bei unklaren Fragestellungen und zum Ausschluss von MDS ergeben sich hier deutliche Vorteile für die klinische Entscheidungsfindung und für weitergehende Diagnostik und Therapiewahl. Frau Dr. C. Bär hat hier die Ergebnisse vorgestellt und konnte zeigen, dass bei unklaren Konstellationen wie Zytopenien die Analyse eines Panels mit bei myeloischen Neoplasien häufig mutierten Gene zu einer deutlichen Verbesserung der klinischen diagnostischen Algorithmen und zu einer Einschränkung der differenzialdiagnostisch in Betracht kommenden Diagnosen führt. Für den Patienten und für den Hämatologen ergibt sich dadurch eine kürzere Zeit bis zur definitiven Diagnose oder Ausschlussdiagnose, die sonst üblichen vielfachen Wiederholungs-Punktionen bei gleicher klinischer Konstellation des Blutbildes können reduziert werden.
Darüber hinaus hat das MLL zusammen mit dem ASH in den letzten zwei Jahren ein Projekt gesteuert, dessen Ergebnis Herr N. Nadarajah beim ASH-Kongress vorgetragen hat. Dabei geht es um die Harmonisierung von molekulargenetischen Befunden, die unter Leitung des MLL und durch gemeinsame Datenauswertung von sechs unterschiedlichen Laboren in Europa und in den USA zu einer gemeinsamen konsentierten Beurteilung von Varianten in Genen geführt hat, die bei hämatologischen Erkrankungen verändert sind. Gemeinsam wurden dabei auch die Algorithmen zur Auswertung und der Laborworkflow zwischen den Laboren diskutiert und abgeglichen. Das Ergebnis wurde im Oktober 2020 zur Verwendung bei der Befundung von hämatologischen Neoplasien auf der ASH Homepage zur Verfügung gestellt und wird jetzt von uns weiter bearbeitet und erweitert.
Ziel der wissenschaftlichen Aktivität des MLL wird es also weiterhin sein, die Diagnostik für unsere Patienten zu verbessern, die Daten im Rahmen von wissenschaftlichen Publikationen und Kooperationen zu teilen und zur Verfügung zu stellen und insbesondere neue technische Möglichkeiten wie das NGS aber eben auch die Anwendung von künstlicher Intelligenz zur Unterstützung der Routinediagnostik im Labor voranzutreiben.
Im Folgenden haben wir alle ASH-Projekte mit MLL-Beteiligung zusammengefasst:
Der Autor

»Sie haben Fragen zum Artikel oder wünschen weitere Informationen? Schreiben Sie mir gerne eine E-Mail.«
Prof. Dr. med. Dr. phil. Torsten Haferlach
Geschäftsführung
Internist, Hämatologe und Onkologe
Stellvertretende Bereichsleitung Zytomorphologie
torsten.haferlach@mll.com